Biología Sintética / Inteligencia Artificial
Desarrollan un modelo de IA que crea proteínas que no existen en la naturaleza
Se habrían necesitado 500 millones de años de evolución para crear naturalmente estos compuestos
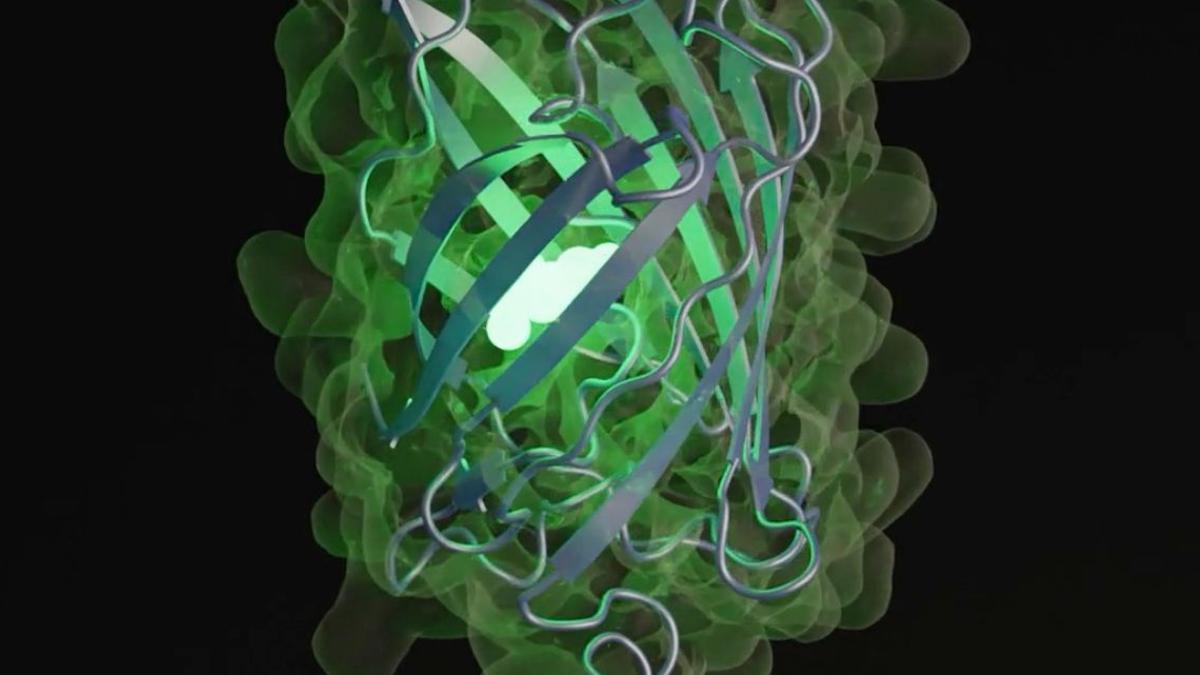
Una representación de esmGFP, una nueva proteína fluorescente verde generada por ESM3 que es diferente a otras proteínas fluorescentes que se encuentran en la naturaleza. / Crédito: EvolutionaryScale.
El modelo ESM3 de Inteligencia Artificial (IA) creado por ex-científicos de Meta puede "escribir" nuevas proteínas desde cero, abriendo grandes posibilidades para el progreso de la biología sintética. Los investigadores desarrollaron con este enfoque una nueva proteína fluorescente que comparte solo el 58% de su secuencia con proteínas fluorescentes naturales.
Investigadores de la startup EvolutionaryScale, creada por ex-científicos de Meta, han presentado en un nuevo estudio publicado en bioRxiv un modelo de Inteligencia Artificial (IA) que puede desarrollar nuevas proteínas que no se encuentran en la naturaleza. Según los especialistas, el proceso artificial “simula” los 500 millones de años de evolución que se necesitan para crear estas estructuras en forma natural.
La biología sintética trabaja en base a la síntesis de biomoléculas o ingeniería de sistemas biológicos con funciones nuevas, que no se encuentran en la naturaleza y que pueden tener múltiples aplicaciones en diversos campos. Ahora, los científicos de EvolutionaryScale utilizaron un nuevo modelo de IA, denominado ESM3, para crear una nueva proteína fluorescente que comparte solo el 58% de su secuencia con las proteínas fluorescentes que se desarrollan naturalmente, según un artículo publicado en Live Science.
Como ChatGPT, pero con proteínas
ESM3 es un modelo de lenguaje grande (LLM) similar al GPT-4 de OpenAI, que impulsa al chatbot ChatGPT. Aunque previamente se lanzó una versión más pequeña de ESM3, el modelo completo incluye 2,78 mil millones de proteínas. Para crear cada proteína, extrajeron información sobre la secuencia, o sea el orden de los componentes básicos de los aminoácidos que forman la proteína, la estructura o forma tridimensional plegada de la proteína y la función, que básicamente es la actividad que lleva adelante el compuesto.
Luego de enmascarar aleatoriamente fragmentos de información sobre estas proteínas, solicitaron a ESM3 que creara los fragmentos faltantes, de la misma manera que ChatGPT genera texto prediciendo la palabra que es más probable que siga en una secuencia discursiva. Este tipo de modelos de IA puede producir un avance sin precedentes en el campo de la biología sintética, al ampliar el horizonte de funciones y aplicaciones de las proteínas naturales.
Un lenguaje por comprender
De acuerdo a una nota de prensa, este tipo de avances busca acercarse de alguna forma a la perfección de la biología, que hasta el momento es la tecnología más avanzada que conocemos, aunque no fue creada por el ser humano. Cada célula de cada organismo de la Tierra tiene de miles a millones de “fábricas moleculares” de proteínas: las mismas son la base de la enfermedad y la salud. Además, muchos fármacos que salvan vidas son proteínas.
Sin embargo, incluso las herramientas informáticas más sofisticadas creadas hasta la fecha apenas rozan la superficie del potencial de la naturaleza: la biología está escrita en un lenguaje que aún no entendemos. Quizás la IA tenga un aporte clave que realizar en el camino por comprender mejor ese lenguaje, con el propósito de utilizarlo para el progreso humano.
Referencia
Simulating 500 million years of evolution with a language model. Thomas Hayes et al. BioRxiv (2024). DOI:https://doi.org/10.1101/2024.07.01.600583
- Supervivientes All Stars': Bosco, cuarto expulsado y Sofía Suescun lidera una inesperada lista de nominados
- El codirector de la cátedra de Begoña Gómez descargó toda la 'responsabilidad' del máster en la mujer de Sánchez
- Vídeo | Un incendio en cinco naves en Polinyà obliga a confinar al núcleo de Santa Perpètua de Mogoda
- Carlos Alcaraz ha ganado cuatro Grand Slams a los 21 años. ¿Cuántos llevaban a su edad Djokovic, Nadal y Federer?
- Mossos cierra su primer macrocontrol de tráfico en toda Catalunya con más de 1.200 denuncias en una sola tarde
- Se extiende hasta el 15 de agosto': el aviso de Mario Picazo sobre lo que nos espera
- Vivir en Osona y que te adjudiquen plaza de maestra a 90 kilómetros: 'Me han dicho que la conciliación no es algo a alegar
- Ikea aterrizará en Granollers el próximo septiembre